
Schrodinger KNIME Workflows 2018-4(药物设计)
附带安装教程- 软件大小:731.88 MB
- 更新日期:2020-04-06 14:40
- 软件语言:简体中文
- 软件类别:辅助设计
- 软件授权:免费版
- 软件官网:待审核
- 适用平台:WinXP, Win7, Win8, Win10, WinAll
- 软件厂商:

软件介绍 人气软件 下载地址
Schrodinger KNIME Workflows 2018-4是一款非常强大的药物设计和材料科学插件套件,同时它还是一款非常专业优秀的分子建模工具,主要是集成在Schrodinger应用系统中;该平台将基于预测性物理的方法与机器学习技术相集成,以加快药物发现,系统的迭代过程旨在在 合成和测定之前加速对硅化学物质的评估和优化;然后,通过额外的计算分析循环,进一步优化来自每一轮实验项目化学过程中最有希望的化合物;与传统方法相比,该平台能够更快,更低成本地发现高质量,新颖的分子,并且成功率会更高;Schrцdinger是计算化学领域的科学领导者,为生命科学和材料研究提供软件解决方案和服务,该程序旨在提供真正满足其客户需求的集成软件解决方案和服务,该程序可以授权世界各地的研究人员通过先进的计算技术来实现改善人类健康和生活质量的目标,这些技术可以改变化学家设计化合物和材料的方式,通过构建和部署突破性的科学软件解决方案并形成合作与伙伴关系,可以帮助科学家加快其研发活动,降低成本并做出原本不可能的新颖发现;需要的用户可以下载体验

新版功能
更快的潜在顾客发现
使用基于物理学的计算来评估类药物分子关键特性的能力,其准确性可与实验实验室测定相媲美,从而有助于优化药物特性,包括药物效价,选择性和生物利用度。
准确的属性预测
使用基于物理学的计算来评估类药物分子关键特性的能力,其准确性可与实验实验室测定相媲美,从而有助于优化药物特性,包括药物效价,选择性和生物利用度。
大规模分子探索
计算思想和探索新颖的,高质量的类药物分子的能力,供发现项目团队考虑,这些对象需要使用经过计算和构造的计算机枚举和生成式机器学习技术,以产生合成上可行的分子。
大规模分子评估
通过将下一代机器学习方法与基于物理的物理方法相结合,能够将我们对关键药物特性的计算扩展到超过十亿个分子的超大型想法集,从而能够更快,更成功地鉴定高质量的候选药物分子技术,以及内部和云计算资源的大规模利用。
集成数据管理和可视化
通过功能强大且用户友好的图形界面,可以生成,访问和分析从与分析数据集成的复杂计算中得出的数据。
软件特色
这些是包含的套件:小分子药物发现套件。
从QSAR到虚拟筛选再到结合亲和力预测,全面的小分子药物发现套件包含用于发现,优化线索的基于片段,配体和结构的药物设计所需的所有工具。
Biologics Suite
这套易于使用的新套件是从头开始设计的,是对建模生物制品,抗体和蛋白质非常重要的所有工具的第一个完整集合。
材料科学套件
这套创新的新套件为基于量子力学的化学系统模拟提供了功能强大的多功能工具,可在特种化学和材料科学中应用对系统进行分析和优化。
Discovery Informatics Suite
下一代用于协作药物设计的平台允许包括医学化学家,生物学家,建模人员和IT专业人员在内的多学科团队实时共享,查看和管理数据。
安装步骤
1、用户可以点击本网站提供的下载路径下载得到对应的程序安装包
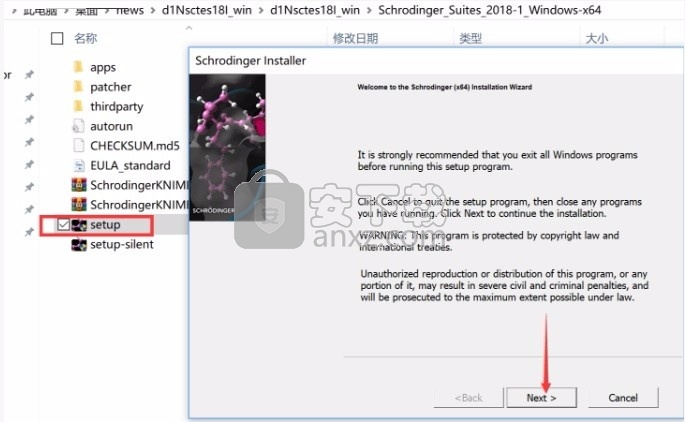
2、只需要使用解压功能将压缩包打开,双击主程序即可进行安装,弹出程序安装界面
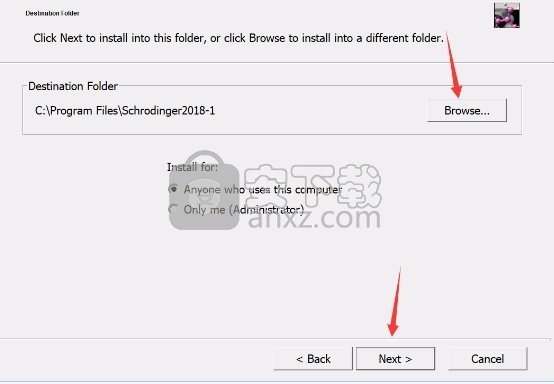
3、可以根据自己的需要点击浏览按钮将应用程序的安装路径进行更改
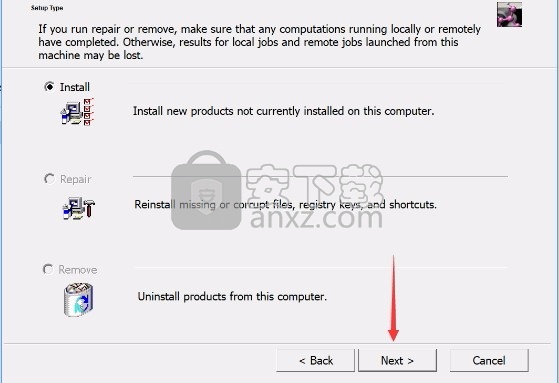
4、弹出以下界面,用户可以直接使用鼠标点击下一步按钮
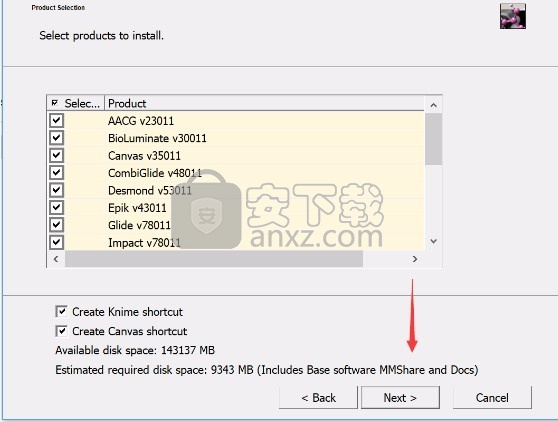
5、弹出以下界面,用户可以直接使用鼠标点击下一步按钮,可以根据您的需要不同的组件进行安装
6、现在准备安装主程序,点击安装按钮开始安装
7、弹出应用程序安装进度条加载界面,只需要等待加载完成即可
8、程序安装完成后,会弹出文件选择界面,根据需要勾选适配文件类型,点next
9、根据提示点击安装,弹出程序安装完成界面,点击完成按钮即可
使用说明
在使用自由能扰动(FEP)方法预测小分子配体与蛋白质的相对结合亲和力方面取得的实质性进展的基础上,我们先前已经表明,在预测突变对蛋白质-蛋白质复合物的结合亲和力。
但是,由于已知的建模扰动(涉及FEP中电荷变化的困难),这些结果仅限于不会改变侧链净电荷的突变。已经提出了各种方法来解决这个问题。
在这里,我们采用化学共水方法研究了FEP计算在先前研究的抗体-gp120系统和其他三种复合物的蛋白质-蛋白质界面处电荷变化突变的功效。
我们在106个案例中实现了1.2 kcal / mol的整体均方根误差,其中涉及通过使用侧链预测和与生物优化项目相关的溶剂可及表面积的简单适用性过滤器选择的净电荷变化。
对于涉及埋藏残基的44个更具挑战性的突变,也获得了合理的结果,尽管准确性不高,在某些情况下,可能需要对局部蛋白质结构进行实质性重组,这可能超出典型FEP模拟的范围。
我们认为,所提出的预测方案将具有足够的效率和准确性,以指导蛋白质工程项目,对于这些项目而言,优化和/或维持高度结合亲和力是关键目标。
一组106个案例中的2 kcal / mol,涉及通过简单的适用性过滤器使用侧链预测和溶剂可及表面积与生物优化项目相关的净电荷变化来选择。
对于涉及埋藏残基的44个更具挑战性的突变,也获得了合理的结果,尽管准确性不高,在某些情况下,可能需要对局部蛋白质结构进行实质性重组,这可能超出典型FEP模拟的范围。
我们认为,所提出的预测方案将具有足够的效率和准确性,以指导蛋白质工程项目,对于这些项目而言,优化和/或维持高度结合亲和力是关键目标。
一组106个案例中的2 kcal / mol,涉及通过简单的适用性过滤器使用侧链预测和溶剂可及表面积与生物优化项目相关的净电荷变化来选择。
对于涉及埋藏残基的44个更具挑战性的突变,也获得了合理的结果,尽管准确性不高,在某些情况下,可能需要对局部蛋白质结构进行实质性重组,这可能超出典型FEP模拟的范围。
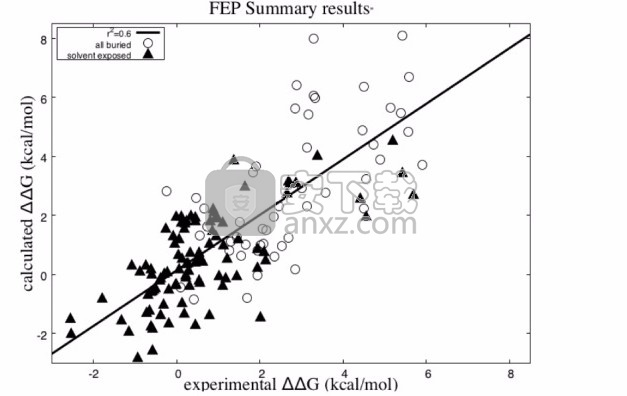
我们认为,所提出的预测方案将具有足够的效率和准确性,以指导蛋白质工程项目,对于这些项目而言,优化和/或维持高度结合亲和力是关键目标。
毫米-GB / SA
分子力学广义玻恩表面积MM分子力学FEP自由能摄动医学博士分子动力学抗体广泛中和抗体RMSE根均方误差SKEMPI突变蛋白相互作用的动力学和能量学结构数据库fSASA分数溶剂可及表面积
残基突变对蛋白质-蛋白质结合亲和力影响的预测是生物分子模拟方法的主要挑战。蛋白质与蛋白质的结合在许多生物学过程中都起着至关重要的作用,包括抗体-抗原结合 [1],γ蛋白质偶联受体信号传导[2],关键分子机器的组装[3]和细胞间识别事件(例如,由钙黏着蛋白介导)[4]。结合亲和力作为突变的函数的计算评估将使这些过程的特异性能够在原子的详细水平上得到理解。此外,健壮且足够准确的方法学可能会对可药用生物制剂(如单克隆抗体和疫苗)的设计产生重大影响。

已经采用了许多方法来预测相对的蛋白质-蛋白质结合亲和力。诸如FoldX之类的工具基于经验测得的蛋白质和蛋白质复合物稳定性数据,使用经过经验训练的能量函数[5] 。诸如分子力学广义玻恩表面积(mm-GB / SA)和分子力学(MM)泊松–玻尔兹曼表面积之类的方法将MM模型与隐式(连续)溶剂分子模型结合使用,从而以更多的计算成本提供了基于物理的方法[6]。还开发了其他半经验方法,这些方法结合了MM方法和根据实验数据优化的其他能量项[7]。可用的此类软件包的示例包括MutaBind [8],它结合了隐式溶剂MM模型中的项和经验能量函数,并通过机器学习将其训练为实验数据,以及BeatMusic,这是一种基于统计的能量函数,其源于已解决的蛋白质结构[9]。自由能扰动(FEP)是一个完全基于物理学的模型,它使用明确表示的水,并具有一系列单独的分子动力学(MD)模拟(“λ窗”),通过该模拟,可变残基的能量权重可以通过在野生型和突变型之间的中间炼金状态,其中每个相邻的λ窗口之间的自由能差是使用扰动展开来计算的,并相加以估计总自由能变化[10]。近年来,现代实现已成为小分子药物发现项目中的宝贵工具
在最近的出版物中,我们对(FEP)方法学的能力进行了大规模测试,以预测与gp120 (HIV- 病毒的病毒尖峰蛋白)结合的抗体中的一系列突变体突变后结合自由能的变化。1 [11] 。抗体与多种gp120蛋白的结合亲和力的优化(导致广泛中和的高效抗体(bNAb))是开发抗体治疗剂以替代HIV感染的小分子疗法的主要目标。进行物理上实际的FEP计算需要建立许多抗体–gp120复合物的同源性模型,并考虑表面聚糖的影响抗体结合到计算中。尽管存在这些挑战,但在55个突变的数据集上,与实验相比,均方根误差(RMSE)为0.84 kcal / mol,这表明在对采样方案进行较小修改的情况下 ,我们之前开发的FEP +方法用于小分子结合亲和力的计算可以成功预测蛋白质突变对蛋白质-蛋白质结合亲和力的影响,接近化学准确性。
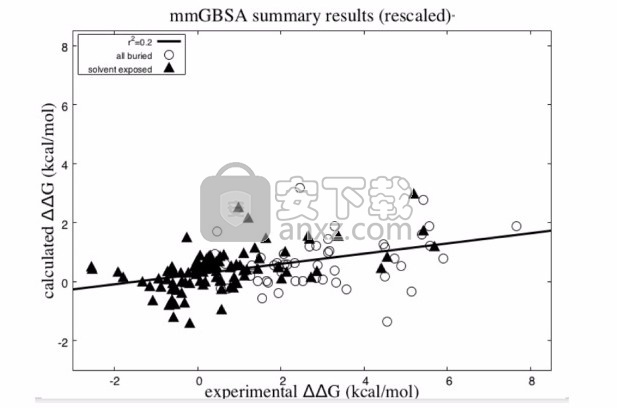
但是,参考文献中检验的测试仪。[11] 完全由不会改变系统净电荷的突变组成;也就是说,从数据集中排除了诸如Ala到Glu之类的电荷变化突变。虽然在施加明确的溶剂自由能的计算以如问题的一些尝试蛋白质折叠存在[13] ,我们故意留出的突变在后一类由于在执行炼金模拟公知的技术困难,其中该系统上的净电荷被更改[14],[15],[16],[17],[18],[19]。我们应用了由Wallace和Shen [15]和Chen 等人首先提出的共化学水FEP方法的变体。[16]。该方法概述如下。参考文献中可以找到更详细的描述以及与其他可用电荷校正方法的比较。[20],重点是小分子/蛋白质FEP计算。
对于先前研究的抗体/ gp120系统,我们已将炼金术离子FEP方法应用于一组点突变,这些突变涉及正常充电的残基(天冬氨酸,谷氨酸,精氨酸或赖氨酸)和在生理条件下正常呈中性的残基。我们排除了脯氨酸的情况,因为脯氨酸的非标准骨架会带来其他技术挑战,而突变型侧链是HIS的情况,因为在没有晶体学证据的情况下确定正确的质子化状态尤其具有挑战性。我们还使用来自突变蛋白动力学和能量学结构数据库的数据,包括了许多其他相互作用的蛋白-蛋白复合物的测试案例。互动(SKEMPI) [21],以便将数据集的大小增加到可以得出初步统计结论的大小。总共考虑了162个点突变,包括19个VRC类抗体与gp-120的结合。
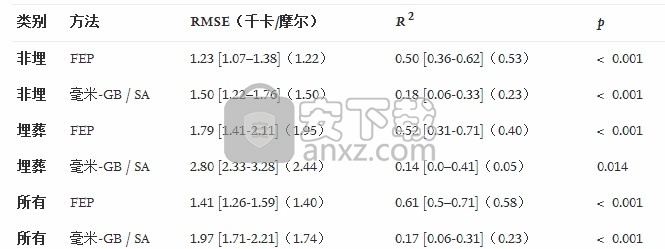
小分子FEP计算通常仅限于预测蛋白构象没有重大变化(例如,激酶中的DFG输入到DFG输出循环运动)的预测。这种运动不太可能在典型的FEP模拟时间尺度上发生。在电荷变化的残基突变的情况下,如果对埋在不适合新靶标残基的环境中的残基进行突变,则很容易引起大量的蛋白质重排。在许多情况下,这种情况将需要更长的模拟时间才能产生完全收敛的结果。幸运的是,它们通常也不会在旨在优化结合亲和力的项目中引起人们的兴趣,因为绝大多数此类突变会使结合更加不利。在下面, 1.2 kcal / mol)。首先,使用隐式溶剂侧链重新预测消除了12种在野生型输入结构中无法实现合理的侧链构象的情况。其次,通过掩埋的野生型残基对具有不同预期电荷状态的残基进行突变的方法是按其分数溶剂可及表面积(fSASA)分类,将SASA标准化为三肽构型的最大SASA [22]。使用10%fSASA的临界值(在该临界值以下,残基侧链被视为完全掩埋)无法通过实验消除任何明显有利的突变,并且留下106例至少部分暴露于溶剂的情况。
本文的结构如下。数据集部分讨论了用于评估FEP计算的各种数据集。在“ 结果和讨论”部分中,给出了所有测试用例的FEP仿真结果,并通过与实验的比较进行了分析。我们使用mm-GB / SA协议对输入结构中的侧链构象进行采样[14],以与更简单(且计算上更便宜)的方法进行比较,并讨论使用经验性foldX方法获得的结果[5]。 ; 这使我们能够质疑FEP是否在增加与大量计算成本相称的价值。在模型和方法中本节中,我们讨论了本文使用的计算方法,包括共炼金水方法,不良拟合和掩埋残渣的表征,以及FEP模拟方法的一些细节,包括使用扩展采样处理氢键和氢键的情况。/或需要断开或形成盐桥。最后,在结论部分中,我们总结了结果并讨论了未来的方向。
数据集
HIV gp120 /抗体数据集
一些感染的患者发展出针对人免疫缺陷病毒的bNAb [1],[24],[25],[26]。这些抗体本身不适合用作治疗剂,但它们为开发更有效的抗体并最终深入了解针对HIV-1的bNAb的进化发展提供了潜在的起点,可将其用于疫苗开发。可靠的计算工具可预测蛋白质残基序列修饰下的亲和力变化,在驱动更有效的抗体设计中可能会很有用,并且能够预测带电和中性侧链之间突变的影响是此类工具的重要组成部分。
在参考文献中 [11] ,我们报告了三种VRC01类抗体(VRC01,VRC03和VRC-PG04)针对中性和电荷变化突变的实验性结合亲和力测量。使用表面等离振子共振测量的实验方案和结果的描述可以在参考文献185中找到。[11],以及补充材料中所有这些突变的实验自由能值(表S1)。
在蛋白质数据库中没有进行实验性结合亲和力测量的抗体/ gp120复合物的结构。然而,具有gp120序列变体的相关结构已经被结晶。在参考文献中 [11] ,我们使用这些结构作为模板来构建同源性模型。参考文献中报道了同源性模型构建方案的细节,用于构建同源性模型的序列比对以及用于验证模型稳定性的MD模拟结果。[11] 。我们还建立了聚糖 根据模板结构的需要,片段是必不可少的任务,因为如果聚糖与所讨论的残基非常接近,它们对突变结合自由能的变化会产生非常显着的影响,并且在考虑的所有情况下都保留在此处。
从SKEMPI数据库中提取的其他测试用例
在带有gp120的VRC01级bNAb的界面中,带电的电荷少于中性残留物;先前报道的丙氨酸扫描集中仅包含19个潜在的电荷变化突变案例[11]。为了建立更广泛的验证数据集来测试FEP电荷变化方法,我们还考虑了来自公共SKEMPI数据库的三种蛋白质-蛋白质复合物的蛋白质残基突变实例[9]。为了选择案例进行分析,推定的电荷变化突变被认为是任何一种末端状态为4-氨基酸侧链(ASP,GLU,LYS和ARG)之一的突变,通常是预期的可以在生理pH值和该组中未包含的任何其他氨基酸侧链上带电。
在选择考虑FEP的案例时,我们旨在组装一组点突变,其中大多数代表我们希望进行的突变类型,该类型与实际生物学优化项目中可能进行的突变最相似,因此我们选择系统此处不存在gp120研究中的结构问题类型(同源模型所需的重要糖基化反应),无法对所测试的特定FEP电荷校正方法进行最佳测试。类似于FEP在小分子药物发现项目中的成功应用[27],[28],我们假设FEP计算将被前瞻性地用于增加亲和力或保持最大可能的亲和力,同时优化其他属性,因此,通过实验对导致亲和力无变化的突变的预测是唯一的。因此,我们要求选择的每个系统都至少具有一个明确的有利突变(ΔΔG≤-0.5 kcal / mol),并且在可以使用多个相似系统的情况下,选择实验上最有利突变的系统。其次,像数据集一样,寻求有利或中性突变的真正优化项目可能不会以丙氨酸突变为主导,因此我们要求选择的每个系统至少要有除丙氨酸以外的氨基酸侧链突变的25%。 ,糖基化,结合的小分子),以最好地将新FEP方案的效果与这些其他挑战区分开。表S1中的SI汇总了被考虑包括在内的具有15个或更多推定电荷变化突变的系统。
选择的系统是复杂的芽孢杆菌RNA酶与芽孢杆菌RNA酶(PDB ID 1BRS ),卵类粘蛋白火鸡第三结构域(OMTKY3)与枯草杆菌蛋白酶嘉士伯(PDB ID 1R0R),和与OMTKY3 灰色链霉菌蛋白酶 B(SGPB)(PDB ID 3SGB)。我们仅从突变氨基酸侧链在物理上不适合参考野生型结构的突变中排除这些突变(更多信息,请参见“ 模型和方法 ”)。剩余的一组被fSASA分为106个溶剂可及的突变和44个掩埋的突变,我们认为只有前者可能对优化复合物的结合亲和力具有实际意义。
最终数据集概述
表1总结了所得的实验数据集。总共包括150个点突变,可以将突变体侧链合理地置于野生型晶体/模型结构中。亲和力变化的动态范围非常大,包括可以强烈稳定结合的突变( 在OMTKY- / SGPB复合物中,低至-2.55 kcal / mol),而对于强烈破坏结合的突变(在非常高的情况下,最高可达7.66 kcal / mol)紧密结合的barnase-barstar复合物)。
使用“ 模型和方法” 部分中概述的协议,我们可以通过FEP模拟从150个点突变中的每个突变获取结合自由能的相对变化的估计值。结果总结在图1中,RMSE和确定系数在表2中给出。图1和表1还提供了mm-GB / SA计算的结果,以进行比较。图3显示了考虑突变的所有位置的位置和野生型电荷。为了使FEP成为有用的方法,它必须大大优于快速近似方法(如mm-GB / SA)(或经验替代方法,如Fold-X [29]),在Ref。中进行了比较。[11])关于预测准确性。
人气软件
-

南方cass 65.9 MB
/简体中文 -

迈迪工具集 211.0 MB
/简体中文 -

origin(函数绘图工具) 88.0 MB
/简体中文 -

OriginLab OriginPro2018中文 493.0 MB
/简体中文 -

探索者TssD2017 417.0 MB
/简体中文 -

mapgis10.3中文(数据收集与管理工具) 168.66 MB
/简体中文 -

刻绘大师绿色版 8.32 MB
/简体中文 -

SigmaPlot 119 MB
/简体中文 -

keyshot6 1024 MB
/简体中文 -

Matlab 2016b 8376 MB
/简体中文


 女娲设计器(GEditor) v3.0.0.1 绿色版
女娲设计器(GEditor) v3.0.0.1 绿色版  iMindQ(思维导图软件) v8.1.2 中文
iMindQ(思维导图软件) v8.1.2 中文  Altair Embed(嵌入式系统开发工具) v2019.01 附带安装教程
Altair Embed(嵌入式系统开发工具) v2019.01 附带安装教程  avizo 2019.1(avizo三维可视化软件) 附安装教程
avizo 2019.1(avizo三维可视化软件) 附安装教程  ChemOffice 2017 附带安装教程
ChemOffice 2017 附带安装教程  绘图助手 v1.0
绘图助手 v1.0 











